도순씨의 코딩일지
딥러닝 :: 데이터 가공하기, 그래프로 표현하기 본문
데이터 가공하기
데이터를 정확하게 파악하기 위해서는 한 번 더 데이터 가공 과정을 거칠 필요가 있다. 우리의 목표는 '당뇨병 발병을 예측하는 것' 이라는 것을 잊지 말아야 한다. 만약 임신 횟수와 당뇨병 발병 확률이 궁금하다면 다음과 같이 계산하면 된다.
|
1
|
print(df[['pregnant', 'class']].groupby(['pregnant'], as_index = False).mean().sort_values(by = 'pregnant', ascending = True))
|
cs |
여기서는 모두 4가지 함수를 사용했다
첫 번째, groupby 함수를 하용하여 'pregnant' 정보를 기준으로 하는 새 그룹을 만들었다.
두 번째, as_index = False는 pregnant 정보 옆에 새로운 index를 만들어 준다.
세 번째, mean 함수를 사용해 평균을 구하고 sort_values 함수를 써서 pregnant 칼럼을 오름차순으로 정리하게끔 설정한다.
결과는 다음과 같다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
|
pregnant class
0 0 0.342342
1 1 0.214815
2 2 0.184466
3 3 0.360000
4 4 0.338235
5 5 0.368421
6 6 0.320000
7 7 0.555556
8 8 0.578947
9 9 0.642857
10 10 0.416667
11 11 0.636364
12 12 0.444444
13 13 0.500000
14 14 1.000000
15 15 1.000000
16 17 1.000000
|
cs |
matplotib를 이용해 그래프로 표현하기
그래프로 표현하지 않으면 이해하기 어려운 데이터들이 많다. matplotlib는 파이썬에서 그래프를 그릴 때 많이 사용되는 라이브러리이다. seaborn 라이브러리는 조금 더 정교하게 그래프를 그리게끔 도와준다.
|
1
2
|
import matplotlib.pyplot as plt
import seaborn as sns
|
cs |
먼저 다음과 같이 패키지를 import 해준다.
|
1
2
|
# 그래프 크기 결정
plt.figure(figsize = (12, 12))
|
cs |
그래프의 크기를 설정해준다.
seaborn 라이브러리 중 각 항목 간의 상관관계를 나타내주는 heatmap 함수를 통해 그래프를 표시해보자. heatmap 함수는 두 항목을 짝을 지은 뒤 각각 어떤 패턴으로 변화하는지를 관찰하는 함수이다. 두 항목이 전혀 다른 패턴으로 변화하고 있으면 0을, 서로 비슷한 패턴으로 변할수록 1과 가까운 값을 출력한다.
|
1
2
3
4
5
|
# 그래프 형식 설정
sns.heatmap(df.corr(), linewidths=0.1, vmax = 0.5, cmap = plt.cm.gist_heat, linecolor = 'white', annot = True)
# 그래프 출력하기
plt.show()
|
cs |
vmax는 색상의 밝기를 조절하는 인자이고, cmap은 matplotlib 색상의 설정값을 불러온다. annot = True는 각 셀에 숫자를 입력하는 것이다.
plot.show()를 이용하여 그래프를 출력하도록 한다.
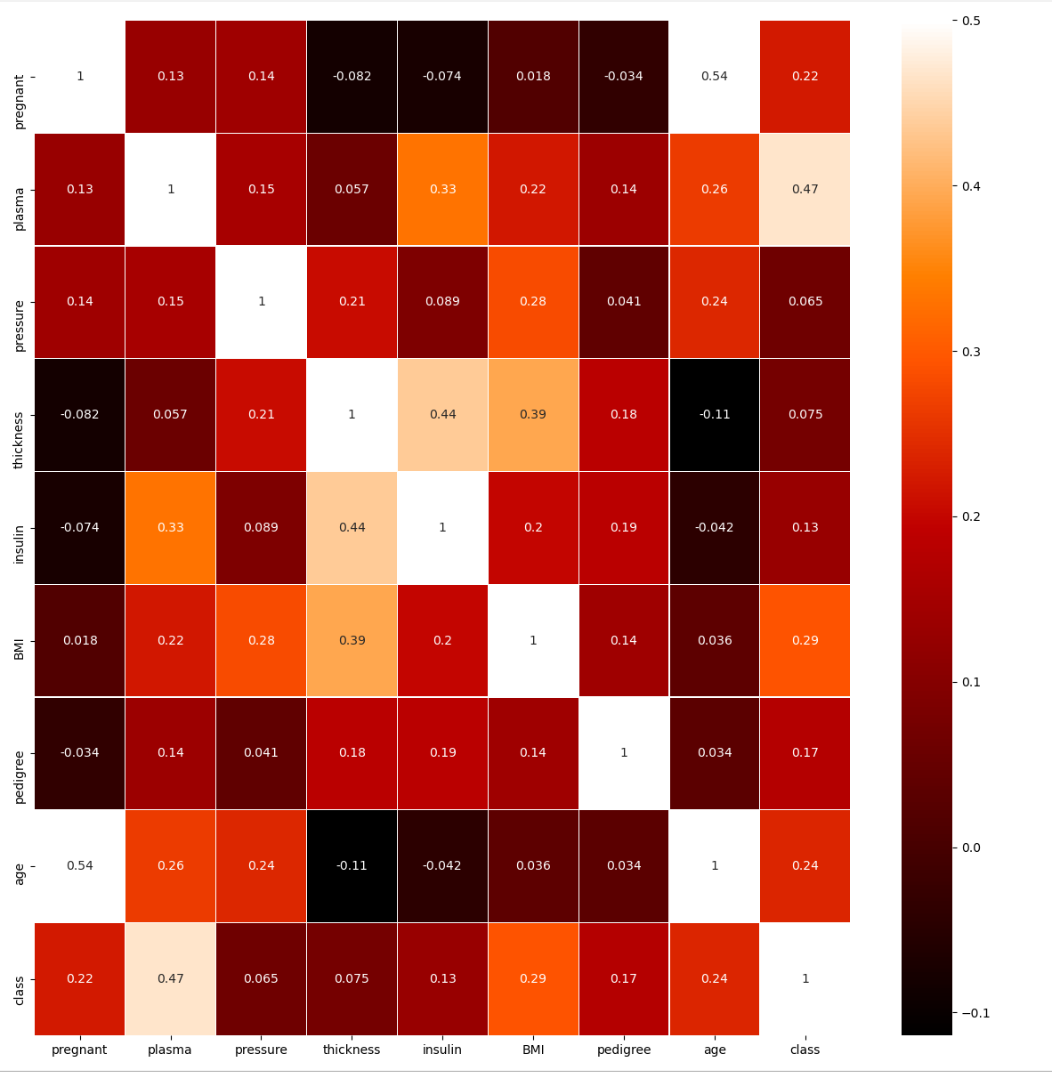
이 중에서도 당뇨병 발병 횟수를 가리키는 class 항목을 자세하게 살펴보도록 하자. 안 쪽에 적혀있는 숫자들을 살펴보면 plasma 항목이 가장 관련이 높다는 것을 알 수 있다(0.47). 그러므로 이 항목이 결론을 만드는데 가장 중요한 역할을 한다는 것을 알 수 있다.
plasma와 class 항목만 추출하여 결과를 살펴보도록 하자.
|
1
2
3
4
|
# plasma, class 항목만 따로 떼어 두 항목 간의 관계를 확인
grid = sns.FacetGrid(df, col = 'class')
grid.map(plt.hist, 'plasma', bins = 10)
plt.show()
|
cs |
FacetGrid는 다중 플롯 그리드를 만들어서 여러가지 쌍 관계를 표현한다. 도화지에 축을 나누는 것과 같다.
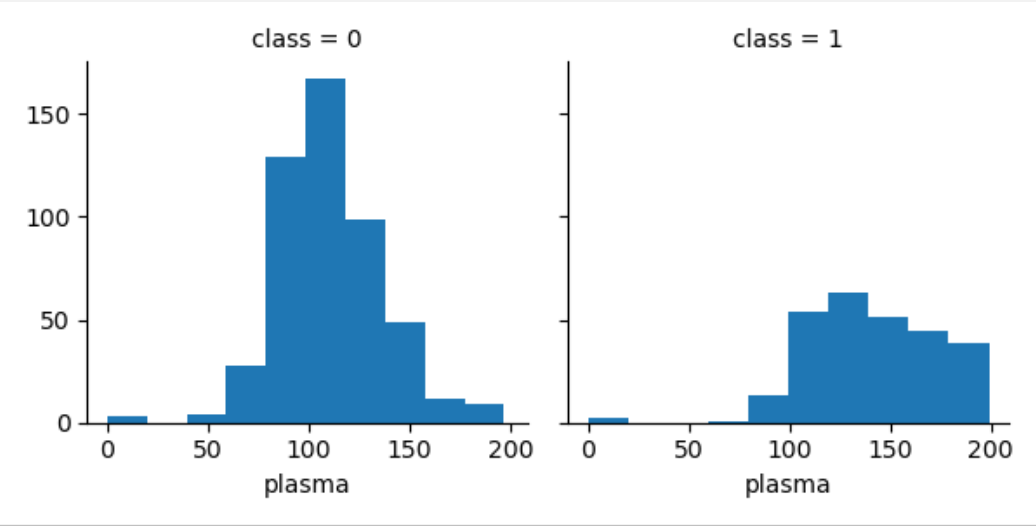
결과는 다음과 같다. 당뇨병이 아닌 환자는 class=0에 해당하고 당뇨병인 환자는 class=1에 해당한다. class=1인 경우 plasma의 수치가 150일 확률이 높다는 것을 알 수 있다.
피마 인디언의 당뇨병 예측 실행
이제 케라스를 이용해 당뇨병 예측을 실행해보자. 먼저 seed값 설정을 살펴보자. seed값을 설정한다는 것은 그 랜덤 테이블 중에서 몇 번째 테이블을 불러와 쓸지를 정하는 것이다.
|
1
2
3
4
|
# seed 값 생성
seed = 0
numpy.random.seed(seed)
tf.random.set_seed(seed)
|
cs |
넘파이 라이브러리를 사용하면서 텐서플로우 기반으로 딥러닝을 구현할 때는 일정한 값을 얻기 위해서 넘파이 seed 값과 텐서플로우 seed 값 모두 설정하여야 한다.
전체 코드와 실행 결과는 다음과 같다.
@ 코드
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
|
import tensorflow as tf
import numpy
from keras.models import Sequential
from keras.layers import Dense
import os
os.environ['KMP_DUPLICATE_LIB_OK']='True'
# seed 값 생성
seed = 0
numpy.random.seed(seed)
tf.random.set_seed(seed)
# 데이터 로드
dataset = numpy.loadtxt("/Users/hansubin/Downloads/006958-master/deeplearning/dataset/pima-indians-diabetes.csv", delimiter = ",")
X = dataset[:, 0:8]
Y = dataset[:,8]
# 모델 생성
model = Sequential()
model.add(Dense(12, input_dim = 8, activation = 'relu'))
model.add(Dense(8, activation = 'relu'))
model.add(Dense(1, activation = 'sigmoid'))
# 모델 컴파일
model.compile(loss = 'binary_crossentropy', optimizer = 'adam', metrics = ['accuracy'])
# 모델 실행
model.fit(X, Y, epochs = 200, batch_size = 10)
# 결과 출력
print("\n Accuracy: %4f" %(model.evaluate(X, Y)[1]))
|
cs |
@ 실행 결과
|
1
|
Accuracy: 0.777344
|
cs |
77.73%의 정확도를 보인다는 것을 알 수 있다.
* 출처
조태호, 모두의 딥러닝(2017), 길벗
https://github.com/gilbutITbook/006958
gilbutITbook/006958
모두의 딥러닝 예제소스. Contribute to gilbutITbook/006958 development by creating an account on GitHub.
github.com
https://rfriend.tistory.com/419
[Python] 히트맵 그리기 (Heatmap by python matplotlib, seaborn, pandas)
이번 포스팅에서는 X축과 Y축에 2개의 범주형 자료의 계급(class)별로 연속형 자료를 집계한 자료를 사용하여, 집계한 값에 비례하여 색깔을 다르게 해서 2차원으로 자료를 시각화하는 히트맵(Heatm
rfriend.tistory.com
https://analytics4everything.tistory.com/66
python 시각화 : Seaborn
Seaborn은 matplotlib 을 기반으로해서, 타이틀 설정 및, 축설정등이 matplotlib 기반으로 한다. 그레프를 조금 더 쉽게 그려주는 페키지이다. 그레프를 통상 관계형, 범주형, 분포, 회귀, 행렬로 나누어�
analytics4everything.tistory.com
'𝐂𝐎𝐌𝐏𝐔𝐓𝐄𝐑 𝐒𝐂𝐈𝐄𝐍𝐂𝐄 > 𝐃𝐄𝐄𝐏 𝐋𝐄𝐀𝐑𝐍𝐈𝐍𝐆' 카테고리의 다른 글
| 딥러닝 :: 과적합 피하기 (0) | 2020.08.11 |
|---|---|
| 딥러닝 :: 다중 분류 문제, 상관도 그래프, 원핫코딩 (0) | 2020.08.10 |
| 딥러닝 :: 모델 설계하기, 교차 엔트로피, 모델 실행하기 (0) | 2020.08.06 |
| 딥러닝 :: 오차 역전파, 활성화 함수, 고급 경사 하강법 (0) | 2020.08.06 |
| 딥러닝 :: [논문분석] 김정미 외 1인, Word2vec을 활용한 RNN기반의 문서 분류에 관한 연구 (0) | 2020.08.06 |


